简介
阿三哥写的文章,哈哈。
这篇文章非常偏应用,问题很具体(虽然我还没搞懂它说类啥),方法也很固定。
里边有很多专业名称,我就不去深究了;哦我查了一下,知道在干啥了,先看流程图吧:
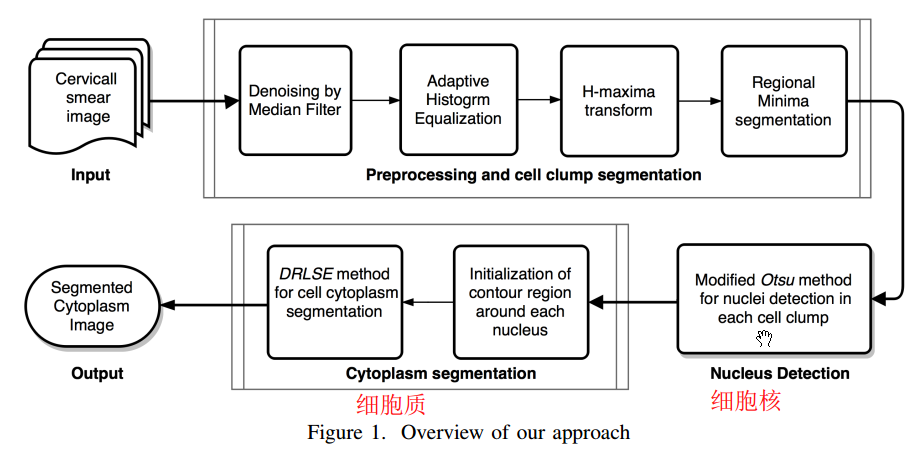
非常中规中矩的cv应用
目的是分割细胞质,不同细胞的细胞质有可能重叠,三哥说就是三步走了:
- 预处理,并把cell的区域分隔开
方法就是先median filter去噪声,然后直方图均衡化以improve对比度,最后采用H-maxima transform:即先suppress intensity image 中的极大值,然后中二值化,如图:
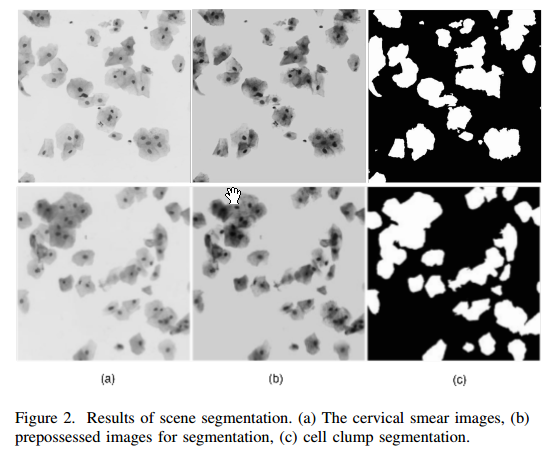
- 细胞核分割
先上图:

中间是Otsus method,最右边是作者搞出来的改进版。Otsus法的输入就是gray-level histogram, Otsus法的定位是:基于聚类的图像阈值化,即得到一个最小化类内variance的阈值。 看来跟聚类是有关系的
三个搞出来的改进版,我没心思看啊。 - 细胞质分割
上图:
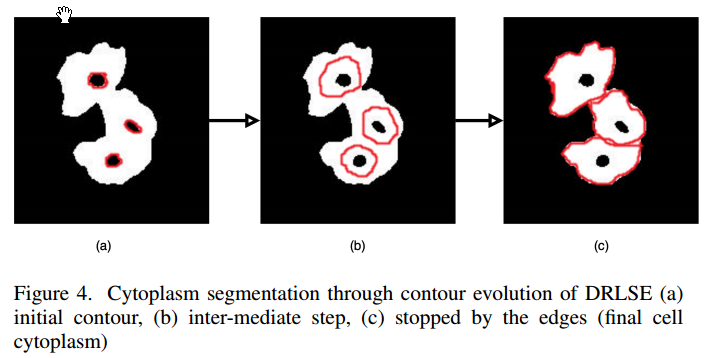
这个用的是现成的方法,就是找到细胞核之后,使用level set evolution就行了,不细究了。
好了,流程就是这样了。
那么第一步河第二步有啥联系啊,我还没搞清楚第二步用的直方图是啥,阿三哥说话不太清晰嘛。
在http://cs.adelaide.edu.au/~zhi/isbi15_challenge/results.html 页面,只有俩队提交了结果,哈哈,我看到它们的paper都很简单吧,都是很基础的cv处理以及简单的intuition(虽然我没看懂哈哈)。
开一下脑洞
-
脑洞1
既然最后目标是分割细胞质,还有可能重叠,那么NPCM不就正好合适么?不过仔细想一下,还是很难的,关键是点咋来?这些像素都用上?肯定得阈值化一下嘛。
恩,可以玩儿一下,反正论文里也提到了数据来源(在实验部分)。
我找到了网站:http://cs.adelaide.edu.au/~zhi/isbi15_challenge/dataset.html -
脑洞2
这次不是玩儿整个流程,而是关注于第二步,即基于clustering的阈值化
总结
这种中规中矩的文章,我只能看到一些新名词吧,可能偶尔会和聚类相关,以后少看吧?